使用Sagemaker Studio 進行ESMFold蛋白質結構預測
使用Sagemaker Studio 進行ESMFold蛋白質結構預測
背景說明
本篇文章提供如何透過Sagemaker Studio,使用Hugging face上的transformer蛋白質模型,簡易測試與部署。
建議瀏覽器
- Chrome
- Edge
Workshop 材料連結
- Workshop 實驗環境連結: <參與workshop時提供>
- 短網址:<參與workshop時提供>
- access code: <參與workshop時提供>
實作課程流程
- 實驗環境準備
- 進入SageMaker Studio
- 執行程式
- 部署endpoint
- 關閉Studio
Workshop 目標
首先,我們會先拿到標準的Herceptin蛋白質結構 source: Protein Data Bank (PDB) 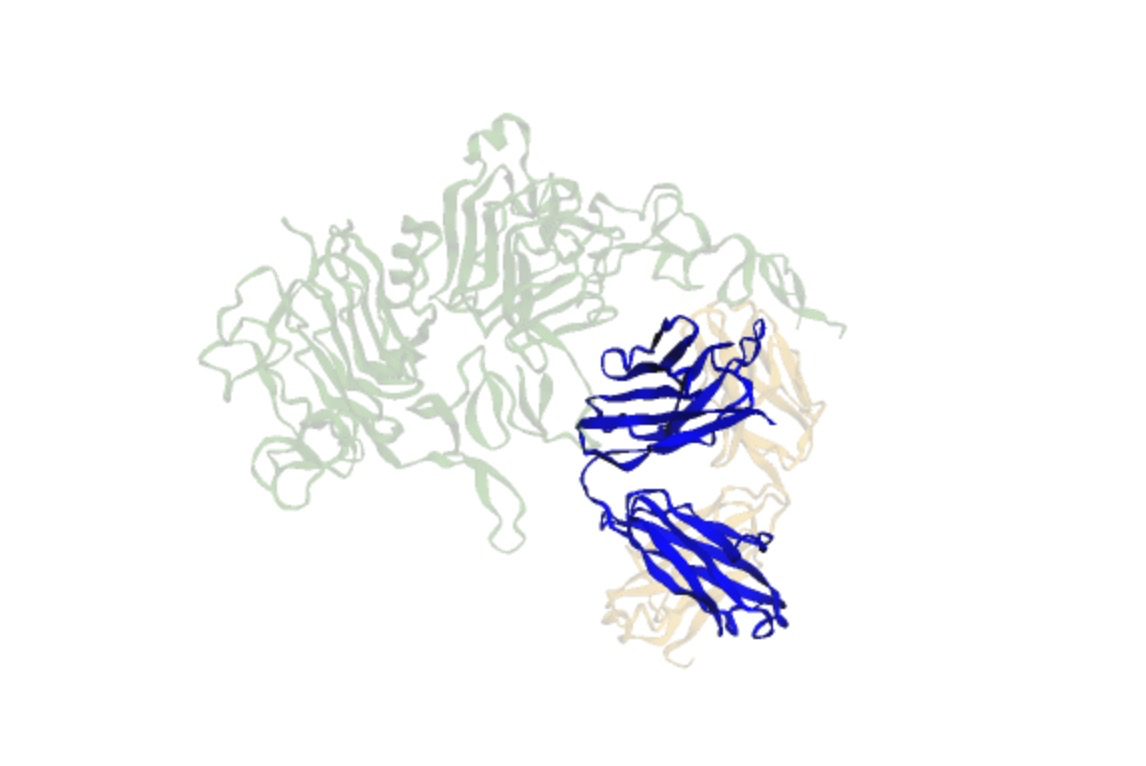 上圖中,橘色的為light chain,藍色的為heavy chain,綠色的為HER2 antigen。
上圖中,橘色的為light chain,藍色的為heavy chain,綠色的為HER2 antigen。 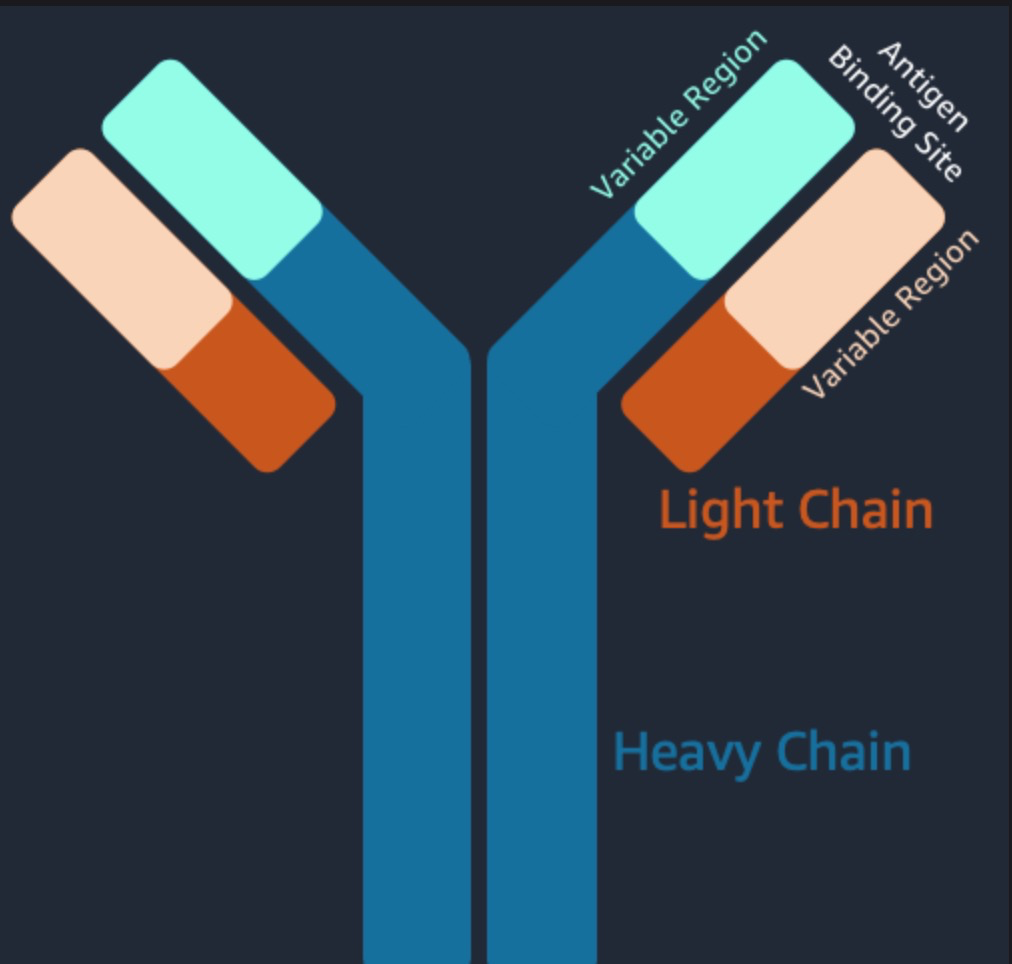
我們嘗試從heavy chain的氨基酸序列預測heavy chain的結構,使用ESMFold模型,並與PDB實驗結構進行比較。我們會使用TM-score作為評分標準。
1. 實驗環境準備
搜尋Sagemaker> Studio > Open Studio 
因為實驗環境限制,如果無法選擇ml.g4dn.xlarge。此時請選則ml.t3.medium
點擊Juypter Lab > Create JupyterLab space > “J1” 
Instance: ml.g4dn.xlarge / ml.t3.medium Storage: 50GB
2. 執行程式
1
git clone https://github.com/aws-samples/aws-healthcare-lifescience-ai-ml-sample-notebooks.git
點擊 aws-healthcare-lifescience-ai-ml-sample-notebooks > workshop > AI_Driven_Protein_Analysis > 1-esmfold-on-sagemaker.ipynb > 選擇Python 3 pykernel 
依照順序執行
- Download and Visualize the Experimentally-Determined Herceptin Protein Structure
- Make an In-Notebook ESMFold Prediction
3. 部署endpoint
因時間關係,本次workshop不執行此部分。此部分會達到的效果為,能夠直接使用API呼叫Sagemaker Endpoint,無需管理底層實作,即可調用ESM2模型進行蛋白質結構設計
4. 關閉Studio
This post is licensed under CC BY 4.0 by the author.